Fechar

O sequenciamento de DNA é o processo realizado para se determinar a sequência exata de nucleotídeos em uma molécula de DNA (ácido desoxirribonucleico). Isso significa que, ao sequenciar um fragmento de DNA, será possível conhecer a sequência em que as quatro bases nucleotídicas (Adenina, Guanina, Citosina e Timina) ocorrem dentro dessa molécula de ácido nucleico.
A sequência nucleotídica é a base para o conhecimento de um gene ou genoma. Ela contém as informações sobre as propriedades hereditárias e bioquímicas da vida. Através dela é possível compreender a construção e estrutura das células e organismos.
O sequenciamento do DNA foi fundamental para que as informações genéticas começassem a ser desvendadas. Por essa razão, é indispensável para a pesquisa biológica.
Os últimos 100 anos testemunharam uma verdadeira revolução no entendimento de como a informação genética é armazenada, usada e transmitida.
Os trabalhos de Mendel, em 1850, foram capazes de demonstrar que as características são transmitidas por hereditariedade ao longo das gerações. Em 1944, o grupo do pesquisador Oswald Avery demonstrou que a transferência de informações entre organismos (no caso, bactérias) é viável e que ocorre pelo material genético, o DNA.
Já em 1953, a estrutura de dupla-hélice do DNA foi desvendada por James Watson e Francis Crick. Isso ajudou no desenvolvimento da biologia molecular e da engenharia genética.
Na década de 1970, Fred Sanger e Walter Gilbert desenvolveram as tecnologias de sequenciamento de DNA. Assim, tornaram possível que a informação genética começasse a ser desvendada.
Até então, a manipulação de genes individuais ou de pequenos trechos do DNA parecia uma tarefa insuperável. O sequenciamento permitiu compreender melhor a estrutura e a função dos ácidos nucleicos. Esse foi o ponto de partida para hoje ser comum isolar e estudar um trecho de DNA.
Frederick Sanger foi responsável por dois dos mais importantes avanços técnicos na área de genética. Primeiro, no início da década de 50, ele desenvolveu um método para determinar a sequência de aminoácidos de um polipeptídio e o fez funcionar no sequenciamento de aminoácidos da insulina.
Este trabalho, finalizado em 1955, forneceu a primeira prova de que os aminoácidos em uma proteína estão presentes em uma sequência constante e geneticamente determinada. Isto deu aos biólogos moleculares a confiança em atacar outros problemas, tais como o código genético.
Então, em 1977, Sanger aperfeiçoou um método rápido de sequenciamento do DNA. Por essa razão, ficou conhecido como método de sequenciamento de Sanger. Abriu assim a porta para a análise da estrutura do gene e sua organização. Ambos os avanços foram valiosos e reconhecidos com prêmios Nobel, em 1958 e 1980.
O método desenvolvido por Sanger foi crucial para desenvolvimento do Projeto internacional do Genoma Humano.
O Projeto Genoma teve como objetivo determinar a sequência de todo o genoma humano. Esse esforço de pesquisa internacional, custando mais de US $ 3 bilhões e levando 13 anos para ser concluído. A primeira sequência completa foi determinada em abril de 2003.
O DNA sequenciado no Projeto Genoma Humano foi obtido a partir de 21 doadores, homens e mulheres, de diferentes grupos étnicos. O resultado possibilitou avaliar muitas informações sobre as diferenças entre os indivíduos de uma mesma espécie.
Esse mapeamento abriu diversas fronteiras e forneceu meios para identificar mutações e doenças genéticas específicas. Pode determinar quais trechos de DNA contêm genes e quais trechos carregam instruções de regulação, ligando ou desligando os genes. Também abriu caminho para uma medicina mais personalizada, permitindo aos cientistas examinar até que ponto a resposta de um paciente a um medicamento é determinada pelo seu perfil genético.
Essas conquistas exigiram o desenvolvimento e aprimoramento de novas tecnologias, com a intenção de melhorar a eficácia dos cuidados de saúde e promover a saúde humana. Claro que a medicina totalmente personalizada ainda está longe de ser uma realidade para todos, mas estes já são os primeiros passos.
O método de sequenciamento de DNA mais usado, até os dias de hoje, é o de Sanger. Também chamado de método didesoxi ou método de terminação de cadeia.
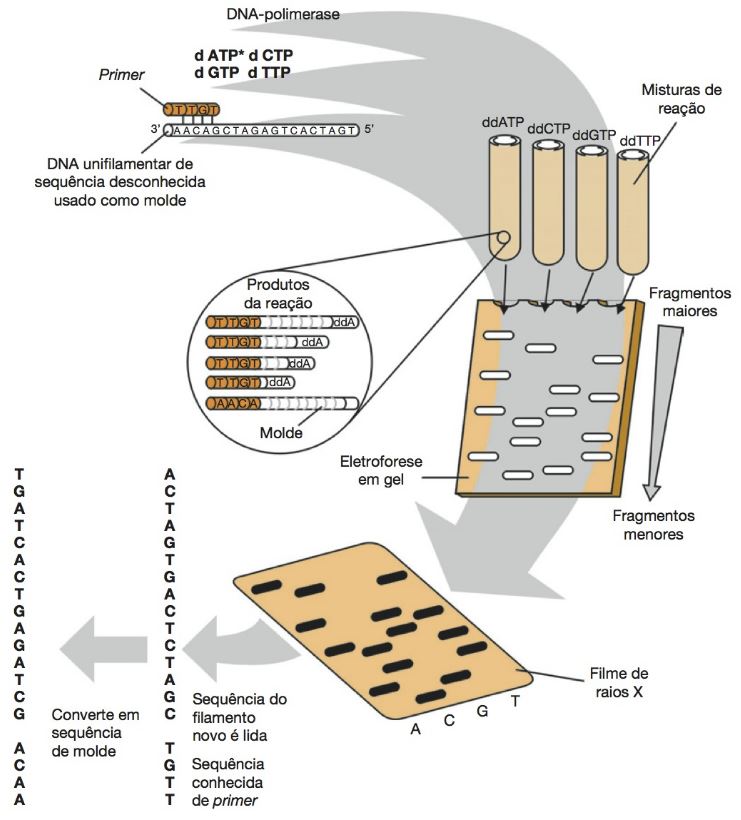
Nesse procedimento, uma molécula de DNA cuja sequência deve ser determinada e? convertida em fitas simples que são utilizadas como molde para sintetizar uma série de fitas complementares. Cada uma dessas fitas termina aleatoriamente em um nucleotídeo específico diferente. A série resultante de fragmentos de DNA e? separada por eletroforese e analisada para revelar a sequência do DNA.
Nesse método são preparados quatro tubos de reação de sequenciamento (A, C, G, T), correspondendo a cada um dos quatro nucleoti?deos. Cada tubo conte?m:
Ocorrem quatro reações diferentes, correspondendo aos quatro didesoxinucleoti?deos marcados (ddATP, ddCTP, ddGTP e ddTTP). Eles terminam a sequência de DNA sempre que são incorporados, ao contrário do desoxinucleoti?deo normal (dATP, dCTP, dGTP e dTTP, correspondentes às bases A, C, G e T, respectivamente).
Isso produz fragmentos com comprimentos variáveis, que podem ser separados por eletroforese.
A posição de cada fragmento no gel e? indicada pela emissão de partículas radioativas do marcador, o que permite a leitura direta da sequência de DNA.
Embora o mesmo método básico seja ainda utilizado, vários aperfeiçoamentos ocorreram. O sequenciamento de DNA automatizado foi um deles.
ORGES-OSÓRIO, Maria Lucena, ROBINSON, Wanyce Miriam. Genética Humana. ArtMed, 2013. pg. 572
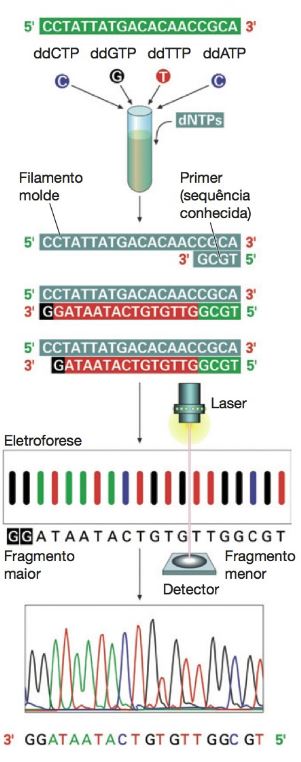
Os avanços técnicos possibilitaram automatizar o sequenciamento, trazendo melhorias para o método de Sanger.
Os equipamentos são capazes de misturar os reagentes, aplica?-los, fazer correr e ler a ordem das bases de nucleotídeos a partir de um gel. O uso de nucleotídeos terminadores de cadeia, marcados com agentes fluorescentes de cores diferentes, facilita esse processo.
As quatro reações de síntese podem ser realizadas no mesmo tubo e os produtos podem ser separados em uma única canaleta de gel por eletroforese capilar. Um detector lê e grava a cor do marcador fluorescente em cada banda a? medida que ela passa. Assim a sequência pode ser analisada.
ORGES-OSÓRIO, Maria Lucena, ROBINSON, Wanyce Miriam. Genética Humana. ArtMed, 2013. pg. 574
Apesar de o método de Sanger ser utilizado até hoje, novas técnicas têm sido desenvolvidas para o sequenciamento de DNA. A mais recente é o sequenciamento de nova geração, também chamada de sequenciamento de segunda geração ou ainda Next-Generation Sequencing (NGS).
Através do NGS é possível realizar o sequenciamento de um genoma inteiro em questão de dias. A sua velocidade, capacidade de gerar dados em larga escala e precisão, a um custo inferior aos métodos tradicionais ganha, a cada dia, mais espaço.
O princípio básico das plataformas de NGS está em sequenciar várias moléculas diferentes (ou vários fragmentos diferentes provenientes do mesmo genoma) em paralelo. Logo, elas diferem fundamentalmente das metodologias de Sanger, em que uma única sequência de DNA, oriunda de um trecho específico, é obtida em cada reação. Isso é possível graças à integração de ferramentas de nanotecnologia, robótica e informática em aparelhos de alto desempenho.
Hoje é possível, portanto, determinar mais rapidamente uma sequência completa de um genoma humano, diferente do período inicial do Projeto Genoma. Os pesquisadores agora são capazes de comparar grandes extensões de DNA (1 milhão de bases ou mais) de indivíduos diferentes, com mais agilidade.
Essas comparações podem gerar uma enorme quantidade de informações sobre o papel da herança na suscetibilidade a doenças e em resposta a influências ambientais. Abrem-se assim novas perspectivas para a medicina baseada nas características genéticas de cada indivíduo, criando um vasto potencial para diagnósticos e terapias.
Da mesma forma, é possível determinar todo o conjunto de microrganismos presente em um ambiente (microbioma) a partir do sequenciamento de todo o DNA ali encontrado. Ou até mesmo comparar as sequências do genoma de diferentes tipos de animais e organismos, fornecendo insights sobre a biologia do desenvolvimento e da evolução.